Une méthode pour comparer le métabolisme des eucaryotes photosynthétiques
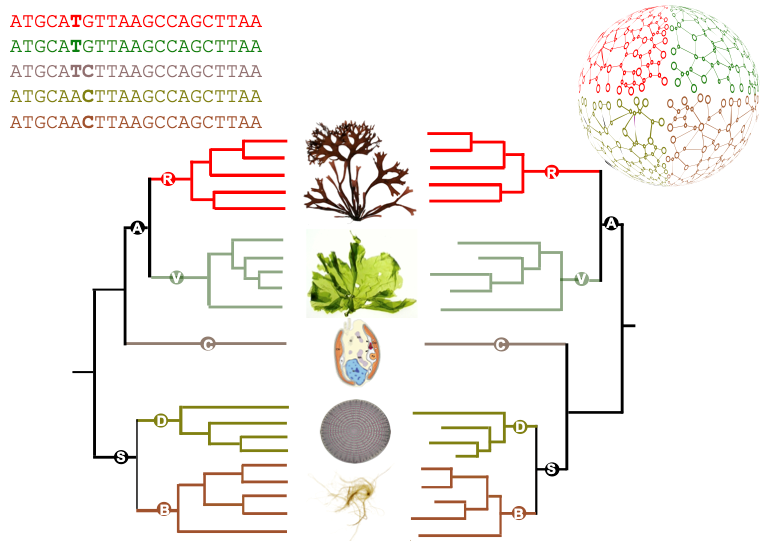
Les réseaux métaboliques à l'échelle du génome permettent de modéliser l'ensemble des transformations chimiques connues au niveau du métabolisme d'un organisme donné. Leur comparaison à large échelle nécessite de s'affranchir des problèmes d'hétérogénéité parmi les génomes disponibles. Dans un article publié dans Genome research, des scientifiques du Laboratoire de biologie intégrative des modèles marins (LBI2M, CNRS/Sorbonne Université), au sein de la Station biologique de Roscoff (CNRS/Sorbonne Université), et de l'Institut de recherche en informatique et systèmes aléatoires (IRISA, CNRS/Université de Rennes) proposent une approche permettant de construire des réseaux de qualité homogène permettant de les comparer dans une perspective évolutive.
Depuis une dizaine d'années, plusieurs cartes du métabolisme des algues ont été reconstruites à partir de données issues de séquençages de génomes. Lors des premières tentatives de comparaison entre ces réseaux métaboliques, les études expliquaient les différences observées entre les cartes métaboliques par l'hétérogénéité des jeux de données issus du séquençage et non par de réelles différences biologiques entre les espèces. La nouvelle approche méthodologique, implémentée dans le logiciel AuCoMe (diffusé sous licence libre), a été développée en utilisant la génomique comparative. Elle permet en s’appuyant sur les connaissances expertes contenues dans les annotations génomiques des espèces les mieux décrites, de déduire les cartes métaboliques des espèces dont les annotations des génomes sont plus rudimentaires – ou incomplètes.
Des gènes manquants ont également pu être identifiés en réalisant des recherches sur les similarités des séquences entre espèces apparentées. Enfin, la méthode intègre aussi une approche pour garantir la fiabilité des prédictions en limitant les biais potentiellement induits par les spécificités d'un génome donné. Cette méthode combine des techniques d'ingénierie des connaissances, de comparaisons de séquences génomiques, et de modélisation de systèmes biologiques pour parvenir à analyser et comparer simultanément le métabolisme de plusieurs espèces eucaryotes, alors que l'état de l'art ne pouvait jusqu'à maintenant que comparer que quelques espèces, et sans pouvoir tenir compte de l'hétérogénéité des génomes.